Známé neznámé DNA sekvenování - Část III
Příliš mnoho signálů – zdvojené píky (= směsné sekvence)
Takže řekněme, že máme dobré a stabilní signály, délka čtení je také v pořádku, takže jsme úspěšně překonali všechny trable zmíněné v prvních dvou částech tohoto textu, ale pořád můžeme být daleko od dobrého výsledku sekvenace. Třetí a poslední část je věnována situaci, že se píky vzájemně překrývají. Sekvence je nečitelná.
V principu mohou nastat tři situace, opět s různými příčinami:
- Zdvojené píky od začátku až do konce
- Zdvojené píky pouze na začátku
- Zdvojené píky pouze na konci
Zdvojené píky od začátku až do konce
Více vazebných míst pro sekvenační primer
Je-li váš templát dostatečně dlouhý a sekvenační primer se váže na dvou různých místech, která jsou obě daleko od konce templátu, je výsledkem směsná sekvence od začátku do konce. Pokud je jedno z těchto vazebných míst sekvenačního primeru poblíž konce PCR produktu, bude výsledkem sekvenace směsná sekvence, která přejde do čisté sekvence, jakmile jedna z těchto sekvenačních reakcí dosáhne konce templátu.
Směs templátů a/nebo primerů
Dva templáty a jeden primer nebo dva primery a jeden templát nebo, v nejhorším případě, několik templátů a několik primerů. Pokud se to stane při sekvenování PCR produktů, musíte zjistit, co se vlastně stalo a postupovat podle toho. S nejvyšší pravděpodobností se jedná o vaši chybu, zřejmě špatně přečištěný templát. Pokud se to stane při sekvenování plazmidové DNA, je dobré si uvědomit, že v dnešní době používané plazmidy jsou skutečně vysoko vysokokopiové. Jelikož účinnost transformace E. coli je rovněž vysoká, může se snadno přihodit, že i když po transformaci získáte jednotlivé kolonie, budou stále obsahovat více různých kopií plazmidu. Doporučujeme vybrat jednotlivou kolonii a na čerstvém agaru z ní ještě jednou získat jednotlivé kolonie.
Zvláštně zdvojené píky (n+1 nebo n-1)
Sekvenační primer je kontaminován n+1 nebo n-1 primerem. Váš primer je ve skutečnosti směs dvou primerů, žádoucího a nežádoucího, který je o jednu bázi kratší nebo naopak delší. Nechte si primer syntetizovat znovu (a zdarma). Není to vaše chyba, je to chyba firmy, která vám ten primer dodala. Můžete zkusit jiného dodavatele.
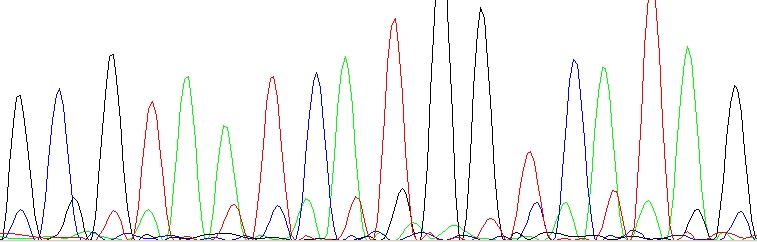
Zdvojené píky pouze na začátku
Primerové dimery
Díky vzájemnému překryvu sekvenačního nebo PCR primeru(ů) vznikly během PCR a/nebo sekvenační reakce krátké nežádoucí produkty, které jsou sekvenovány spolu se žádoucím produktem. Buď je nutné navrhnout nový primer(y) nebo zvolit správný způsob přečištění (viz výše).
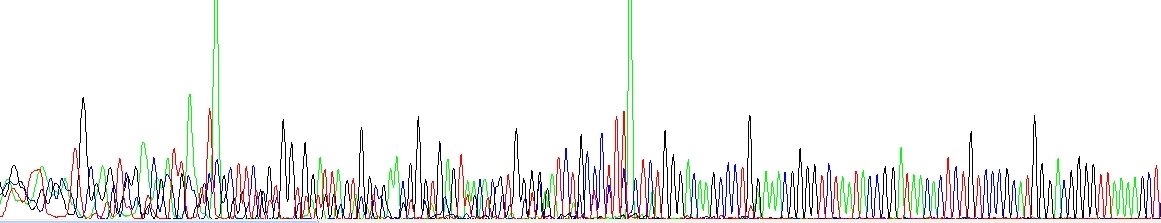
Primerové dimery a/nebo další krátké nespecifické produkty. Povšimněte si velkých zelených píků na konci každého nespecifického produktu, vzniklých přidáním A Taq polymerázou na 3'-konec – je to poměrně symptomatické.
Více vazebných míst pro sekvenační primer – Viz výše
Směs templátů a/nebo primerů – Opět viz výše
Zdvojené píky pouze na konci
Za homopolymerním úsekem nebo repeticí
Toto je poměrně specifický případ, získané elektroferogramy jsou velmi charakteristické. Kdysi jsem od někoho slyšel pojmenování “běžící ježci” :-D což je velmi příhodné.
Taq DNA polymeráza používaná při PCR a/nebo DNA sekvenování není příliš procesivní. To znamená, že po polymerizaci krátkého úseku DNA (řekněme průměrně nějakých 35 nukleotidů) dojde k disociaci enzymu od templátu a 3’ konec vznikajícího řetězce se tedy z templátu uvolní. (Naproti tomu např. T7 DNA polymeráza nasyntetizuje nějakých 1,000 nukleotidů a replikativní DNA polymerázy milióny bází než dojde k jejich disociaci od templátu. Při cyklickém sekvenování se však tyto enzymy nedají použít.) Pokud k tomu dojde v nějaké homopolymerní oblasti a 3’ konec následně nasedne zpět na templát, může nasednout přesně tam kde má, ale také o bázi vpřed nebo vzad. Při následné extenzi produktu za homopolymerním úsekem pak dostaneme vlastně směs produktů lišících se o bázi, o dvě i víc ve srovnání s templátem. Následná sekvenační elektroforéza tuto směs rozdělí, takže až k homopolymeru máme pěknou a čistou sekvenci a za ním máme směs. Jak velká směs to bude záleží primárně na délce homopolymerní oblasti.
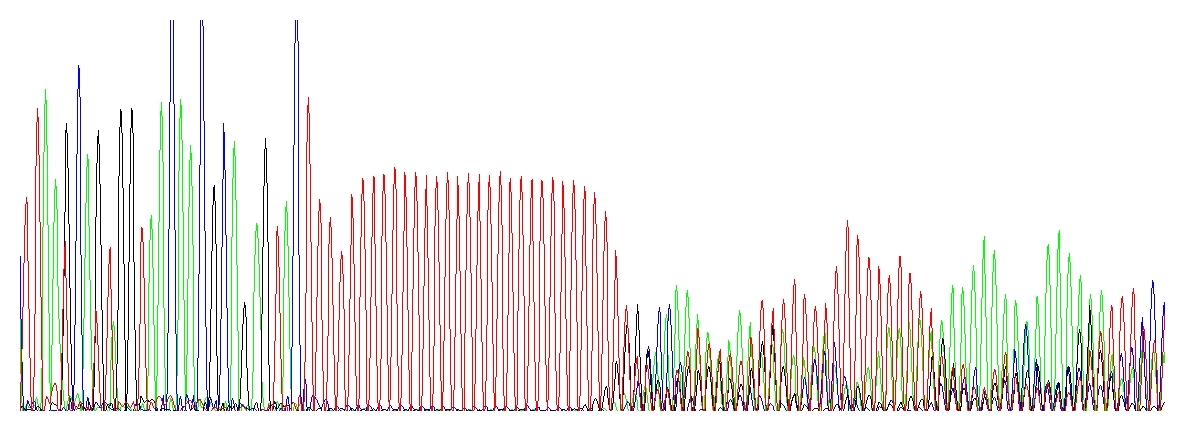 „Běžící ježci“ uhánějí za červeným polyT úsekem
„Běžící ježci“ uhánějí za červeným polyT úsekem
Vyřešit tento problém uspokojivě je opět docela výzva, podobně jako v případě vlásenek. Máme-li takovou sekvenci v plazmidu nebo v restrikčním fragmentu z plazmidu, můžeme úspěšně pročíst skrz třeba až 15 opakovaných stejných nukleotidů než se problém objeví. Jedná-li se o PCR produkt, problém se vyskytne i na sekvenci 8–10 opakovaných nukleotidů a někdy i kratší, protože ke skluzu polymerázy dojde nejen při sekvenační reakci, ale i při předcházející PCR. Řešení tedy spočívá ve vynechání PCR kroku, klonování fragmentu do plazmidu a sekvenování v plazmidu. Další možností je navrhnout primer, který obsahuje homopolymerní oblast. Jedná-li se řekněme o poly A úsek, bude primer oligo dT s C, A nebo G jako 3’-koncovým nukleotidem a obdobně pro reverzní řetězec. Pomocí těchto primerů sekvenujeme ve směru od homopolymerní oblasti a pomocí další dvojice primerů navržených okolo této oblasti směrem do ní. Tím lze většinou získat uspokojivé čtení, ale zjistit přesný počet nukleotidů v homopolymerní oblasti bude pravděpodobně stále značný problém.
Za čistou sekvencí (žádné homopolymery ani repetice) – Inzerce nebo delece
Sekvence je čistá a čitelná a najednou je zdvojená. Templát je opět směs zřejmě dvou templátů, z nichž jeden obsahuje např. bázi navíc. Lze vyřešit sekvenováním v obou směrech.
Závěrem bych chtěl upozornit, že informace obsažené v tomto textu jsou součástí našich sekvenačních seminářů. Organizujeme je po dohodě s vámi, ve vaší instituci či na vašem oddělení, s cílem vyrovnat rozdíly ve znalostech jejich účastníků a doporučit správné pracovní postupy. Časový rozsah i náplň jsou velmi variabilní, počínaje stručnou prezentací zaměřenou na základní informace a konče velmi podrobným několikadenním školením. Více informací zde.