Přečištění PCR produktů – srovnání kolonkových souprav různých výrobců a naše tipy
Upozornění:
Cílem tohoto testu bylo zjistit, zda a jaký vliv mají soupravy pro přečišťování PCR produktů na kvalitu výstupů Sangerova sekvenování v naší laboratoři. Níže učiněné závěry a doporučení nejsou obecně platné a doporučujeme každému, aby si obdobný test provedl za použití vlastních vzorků sám.
Proč jsme testovali
Při sekvenování PCR produktů někdy dochází k tomu, že se sekvence PCR produktu nepřečte celá:
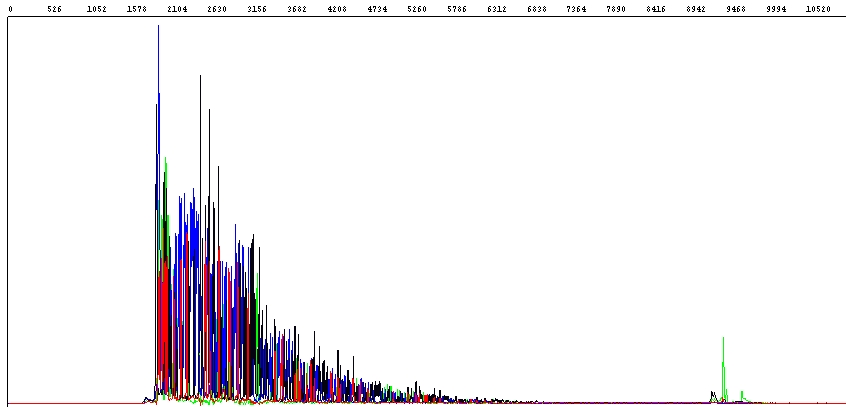
Jak vidíte v hrubých datech na obrázku výše, fluorescenční signál je zpočátku velmi dobrý, ale prudce klesá a zhruba polovina sekvence PCR produktu je nečitelná (konec PCR produktu je vidět jako výrazný zelený pík vpravo).
Typickou příčinou tohoto problému je přítomnost různých inhibitorů v templátech sekvenační reakce. Může se jednat o inhibitory obsažené v původních biologických materiálech (tkáň, krev, půda apod.), které nebyly při izolaci DNA odstraněny, nebo inhibitory, které se k DNA dostanou při její izolaci nebo přečišťování (zbytky vazebných a/nebo promývacích pufrů – různé soli, detergenty, alkoholy).
Metody izolace genomické DNA jsou poměrně variabilní a závisí na typu vstupního materiálu. Pro potřeby Sangerova sekvenování je často typickým dalším krokem PCR amplifikace získané genomické DNA a přečištění PCR produktu, což již je krok, který je relativně standardizovaný. PCR produkty lze čistit enzymaticky, vyřezávat z gelů případně přečistit na kolonkách apod. Na tento poslední způsob – přečišťování PCR produktů na centrifugačních kolonkách jsme se v našem testu zaměřili. Naším cílem bylo zjistit, jaký mají soupravy na přečišťování PCR produktů od různých výrobců vliv na kvalitu (čistotu) získaného DNA templátu a následnou sekvenaci.
Jak jsme testovali
V dnešní době si na trhu můžete vybrat z nepřeberného množství souprav na přečišťování PCR produktů, které se však v řadě detailů liší. Mezi hlavní odlišnosti patří:
- design kolonky (úzké/široké hrdlo silika-membrány)
- doba centrifugace
- RCF
- počet promytí promývacím pufrem
- přidaný krok dodatečné centrifugace před elucí
- doba eluce
- objem elučního pufru
Pro účely tohoto testu jsme vybrali čtyři běžně používané soupravy na přečišťování PCR produktů:
- NucleoSpin® Gel and PCR Clean-up, 740609.10, Macherey-Nagel, protocol version 02/2017/Rev. 04, dále vedeno pod názvem Macherey-Nagel
- Monarch® PCR & DNA Cleanup Kit (5 μg), T1030G, New England Biolabs, protocol version 1.1-10.14, dále vedeno pod názvem NEB
- QIAquick PCR Purification Kit, 28104, QIAGEN, protocol version 07/2018, dále vedeno pod názvem Qiagen
- DNA Clean & Concentrator-5 Kit™, D4003S, ZYMO RESEARCH, protocol version 1.2.1., dále vedeno pod názvem Zymo
Srovnání hlavních parametrů protokolů vybraných souprav (tabulka A):
| Souprava | RCF | Doba centrifugace | Počet promytí | Objem promývacího pufru (ul) | Centrifugace navíc před elucí | Doba inkubace s elučním pufrem | Objem elučního pufru (ul) |
|
Macherey-Nagel
|
11000
|
30 sec (1 min po eluci)
|
2x
|
700
|
1 min
|
1 min
|
15-30
|
|
NEB
|
16000
|
1 min
|
2x
|
200
|
volitelně
|
1 min
|
>=6
|
|
Qiagen
|
17900
|
30-60 sec (1 min po eluci)
|
1x
|
750
|
1 min
|
1 min
|
30-50
|
|
Zymo
|
>=10000
|
30 sec
|
2x
|
200
|
ne
|
1 min
|
>=6
|
Pro potřeby našeho testu jsme použili takto upravené protokoly (tabulka B):
| Souprava | RCF | Doba centrifugace | Počet promytí | Objem promývacího pufru (ul) | Centrifugace navíc před elucí | Doba inkubace s elučním pufrem | Objem elučního pufru (ul) |
|
Macherey-Nagel
|
Dle protokolu
|
20
|
|||||
|
NEB
|
Dle protokolu
|
ne
|
Dle protokolu
|
15
|
|||
|
Qiagen
|
16000*
|
1 min
|
Dle protokolu
|
30
|
|||
|
Zymo
|
16000
|
Dle protokolu
|
15
|
||||
* Max. hodnota RCF námi použité centrifugy je 16000.
Stejné PCR produkty (o různých délkách od 150 do 750 bp) byly vždy přečištěny různými soupravami dle protokolů uvedených v tabulce B. Na kolonky bylo nanášeno vždy stejné množství PCR produktu. Kvantitativní výtěžek (měřeno pomocí Qubit 4 Fluorometer) byl přepočítán tak, aby do sekvenační reakce vstupovalo vždy stejné množství DNA. Možné rozdíly v kvalitě sekvenačních dat tedy nemohly být způsobeny různým množstvím templátu v sekvenační reakci. To ovšem neplatí o vstupním objemu templátu – u kitů s nižším výtěžkem a/nebo vyšším elučním objemem byl do sekvenační reakce přirozeně použit vyšší objem templátu (a tedy i potenciálně více inhibitorů). Přečištěné PCR produkty byly jednosměrně sekvenovány protokolem StandardSeq. Kvalita sekvenačních dat byla hodnocena prostým náhledem na hrubá data a elektroferogramy. Hodnocení provedli tři operátoři nezávisle na sobě v programu Sequencing Analysis (Applied Biosystems).
Výsledky:
Při porovnání získaných dat jsme zjistili významné rozdíly. Níže jsme vybrali několik příkladů, kde jsou tyto rozdíly jasně viditelné. Všechny obrázky ukazují výsledek sekvenační analýzy totožného PCR produktu (o délce asi 600 bp) přečištěného různými soupravami.
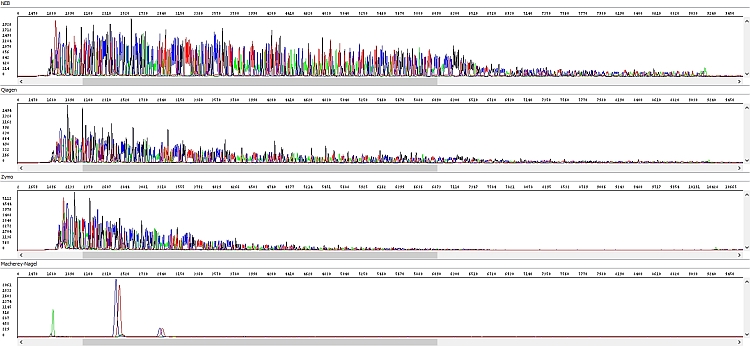
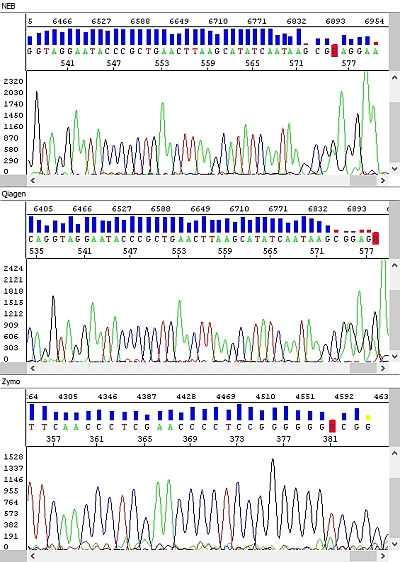
Obrázek 2 - Hrubá data - okna shora dolů: NEB, Qiagen, Zymo, Macherey-Nagel:
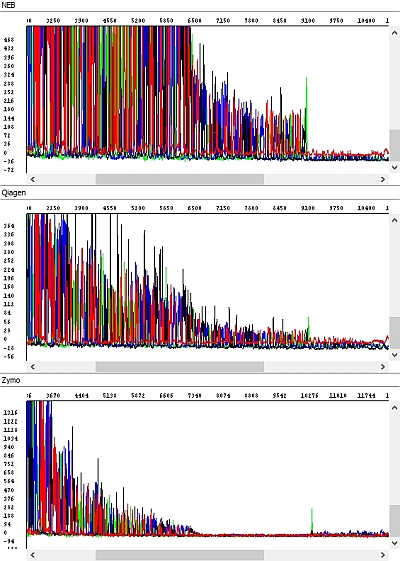
Obrázek 3 - Hrubá data, detail, náhled na konec sekvence PCR produktů - okna shora dolů: NEB, Qiagen, Zymo:
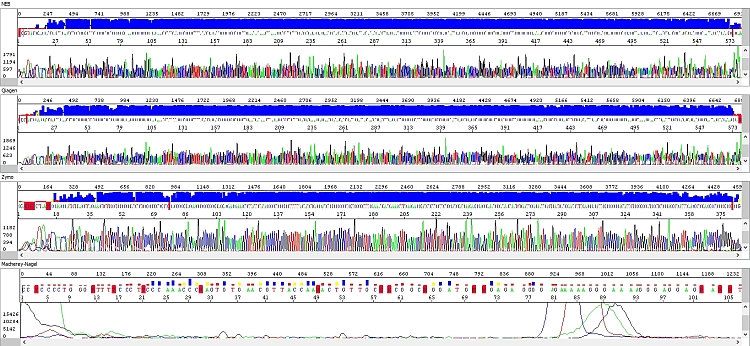
Obrázek 4 - Elektroferogramy - okna shora dolů: NEB (délka čtení QV20 559 bp), Qiagen (délka čtení QV20 559 bp), Zymo (délka čtení QV20 361 bp), Macherey-Nagel (délka čtení QV20 18):
Obrázek 5 - Elektroferogramy, detail, náhled na konec sekvence PCR produktů - okna shora dolů: NEB, Qiagen, Zymo:
Komentář k výsledkům
PCR produkty přečištěné soupravou NEB byly vždy bez problému, s velmi kvalitními daty po celé délce sekvenačního čtení (případný pokles fluorescenčního signálu souvisel spíše s charakterem templátu – kombinace jednotlivých bází, sekundární struktury apod.)
Podobné výsledky měla souprava Qiagen a ačkoliv u delších fragmentů signál rychleji klesal (viz hrubá data), byl stále pro sekvenační analýzu zcela dostačující. Nicméně se domníváme, že v případě nižší než optimální koncentrace PCR produktu by se u kitu Qiagen nemusel PCR produkt dočíst až do konce, k čemuž by u kitu NEB pravděpodobně došlo jen u PCR produktu s opravdu nízkou koncentrací. Vliv také může mít požadavek vyššího objemu elučního pufru a výše zmíněných důsledků z toho plynoucích. Každopádně z hlediska našich požadavků na koncentraci templátů je kit Qiagen bez problému použitelný.
U soupravy Zymo jsme zaznamenali výrazné zhoršení kvality sekvenačních dat – délka sekvenačního čtení byla zhruba poloviční a v hrubých datech je jasně vidět absence detekovatelných píků ke konci sekvence s výjimkou výraznějšího adeninu na 3´konci PCR produktu.
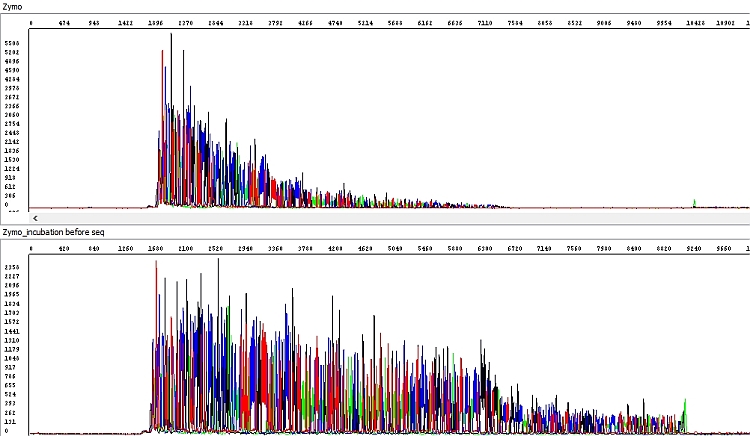
PCR produkt přečištěný soupravou Zymo jsme proto následně před použitím v sekvenační reakci inkubovali 60 min při pokojové teplotě s otevřeným víčkem zkumavky a výsledek byl o poznání lepší, v podstatě perfektní (viz níže). Problémem prvního vzorku byla pravděpodobně přítomnost zbytkového ethanolu z promývacího pufru, který nebyl standardním postupem výrobce odstraněn. Situaci by zřejmě řešila dodatečná centrifugace před elucí (1 min) jako např. u soupravy Qiagen, nicméně pro soupravu Zymo není výrobcem předepsána. My ji každopádně doporučujeme, v tomto testu nebyla provedena.
Obrázek 6 - Hrubá data získaná sekvenací PCR produktu přečištěného soupravou Zymo dle protokolu uvedeného v tabulce B (horní okno, délka čtení QV20 361 bp) a s dodatečnou inkubací kolonky (dolní okno, délka čtení QV20 560 bp):
Nejhůře dopadla souprava Macherey-Nagel. Některé analýzy nevycházely vůbec, a to ani po stejné modifikaci protokolu jako v předchozím případě, výsledkem byla absolutně „mrtvá“ sekvenační reakce. K mírnému zlepšení došlo po inkubaci samotné kolonky (70°C, 5 min) před nanesením elučního pufru, kdy mohly být kompletně odpařeny nežádoucí těkavé organické složky. Výsledek analýzy byl však stále neuspokojivý. Je rovněž nutno podotknout, že u většiny testovaných PCR produktů měla souprava Macherey-Nagel nižší výtěžek izolace. V kombinaci s vyšším objemem elučního pufru (oproti soupravám NEB a Zymo) mohl být špatný výsledek způsoben i tím, že ačkoliv do sekvenační reakce šlo kvantitativně stejné množství templátu, jeho objem byl vyšší. Souprava Macherey-Nagel tedy nemusí být ideální pro sekvenování, ale je třeba dostačující pro jiné aplikace.
Obrázek 7 - Hrubá data získaná sekvenací PCR produktu přečištěného soupravou Macherey-Nagel dle protokolu uvedeného v tabulce B (horní okno) a s dodatečnou inkubací kolonky (dolní okno):

Souhrn a naše doporučení
Pokud jde o kvalitu DNA templátu pro sekvenační reakci, naše zkušenosti jednoznačně ukazují, že výběr purifikační soupravy je zásadní. Pakliže Vaše sekvenační reakce, kde jako templát používáte PCR produkty, nevycházejí optimálně, můžeme kromě změny výrobce purifikační soupravy zmínit následující doporučení či modifikace protokolů:
- Veškerý ethanol z promývacích pufrů musí být ze silika-membrány dokonale odstraněn - dodatečnou centrifugací před elucí, pootočením vnitřní zkumavky kolonky během opakované centrifugace, centrifugací kolonky s otevřeným víčkem umožňující lepší průchod vzduchu přes membránu, dokonalým vysušením membrány před nanesením elučního pufru (inkubace 3-5min při RT), „vyvětráním“ finálního vzorku
- Ethanol v promývacích pufrech musí být čistý
- Pro odstranění jiných inhibitorů (např. soli, detergenty, agaróza) lze provést i několikanásobné promytí silika-membrány promývacími pufry (což však může být na úkor žádaného výtěžku a rychlé spotřeby reagencií purifikační soupravy)
- Centrifugaci s promývacím pufrem lze provést až např. po 2-5 min po jeho nanesení na membránu (dochází k lepšímu rozpuštění a odstranění solí)
- Rozvaha optimálního objemu elučního pufru – samozřejmě platí, že čím menší objem zvolíme, tím získáme vyšší koncentraci finálního vzorku, ale také potenciálních inhibitorů!
Závěrem doporučujeme náš sekvenační protokol CleanSeq, který zahrnuje i přečištění PCR produktů, takže se problematikou popsanou výše nemusíte vůbec zabývat :-)
Sanger lab, info@seqme.eu